El término «metagenómica» fue utilizado por Jo Handelsman, Clardy Jon, Robert M. y Goodman, apareciendo por primera vez en 1998.
La palabra hace referencia al estudio de una multitud de genomas que provienen de distintos organismos procedentes de un mismo entorno. Estos genomas se estudian como si se tratase de un solo organismo.
Otros investigadores definen la metagenómica como: «el uso de técnicas genómicas avanzadas para el estudio de comunidades de microorganismos directamente en su entorno natural, eludiendo la necesidad de aislamiento y cultivo en laboratorio de especies individuales. »

Sin embargo estos estudios tienen un valor principalmente taxonómico y nos dan una visión muy general.
Pero, ¿qué tiene que ver la metagenómica con las plantas de arroz?
El arroz, al igual que muchos otros organismos posee bacterias que viven en su interior realizando todo tipo de funciones, estos microorganismos tienen bastantes aplicaciones económicas, sin embargo su estudio y aprovechamiento no es nada sencillo.
El lugar suele ser el primero de los problemas, identificar los microorganismos que viven, ya sea en una raíz o en un manantial no es tarea sencilla, no se puede identificar visualmente cada especie, contar u observar interacciones al primer vistazo. Necesitamos tomar muestras y buscar crecimiento en el laboratorio, sin embargo no conocemos los requerimientos de todos los microorganismos, por lo que una enorme cantidad de ellos nunca llegan a crecer en las placas. Además los medios de cultivo no son para nada iguales a las condiciones naturales de donde los extraemos, por lo que sus respuestas pueden ser distintas.
Por si esto no fuese poco, en el caso de las raíces encontramos un problema añadido, y es que encontramos tres comunidades claramente diferenciadas.
La primera es la llamada: ectorrizosfera, que comprende las zonas de suelo que rodean a la raíz y que se encuentran bajo la influencia de la misma. No hay que olvidar que cerca del 40% del carbono fijado por la planta puede ser excretado por las raíces en forma de nutrientes. Estos nutrientes atraen a multitud de microorganismos que establecen relaciones con la planta.
La segunda zona se llama rizoplano, y comprende a los microorganismos que viven pegados a la raíz. Por último tenemos a los organismos endofíticos o endorrizosféricos, que son todos aquellos que habitan dentro de la propia raíz. En el interior estos microorganismos establecen relaciones simbióticas de una enorme importancia.
Son estos últimos los que especialmente nos interesan, aunque ya podréis imaginar la dificultad que puede entrañar tomar muestras del interior de una raíz buscando distintos tipos de bacterias, bacterias que quizás luego no sean capaces de crecer en el laboratorio, fuera de la raíz.
Por ello una buena aproximación para el estudio de las comunidades endofíticas consiste en realizar análisis metagenómicos.
¿Por qué el arroz?
Con el aumento en la demanda de alimentos necesitamos conocer mejor que nunca qué microorganismos afectan y cómo lo hacen en distintas plantas, en especial sobre aquellas de gran importancia alimentaria.
El arroz es la base en la nutrición de una importante parte de la población, según algunos datos de la FAO en 2010 se llegaron a producir 454 millones de toneladas de arroz de las que 389 se dedicaron al consumo humano.
La producción de arroz irrigado consume, sólo en Asia, el 40% de las reservas hídricas. Además de consumir el 10% de la producción mundial de fertilizantes nitrogenados.
¿Cómo podrían las bacterias cambiar esto?
Las comunidades endofíticas que viven en tejidos sanos de las plantas son parte fundamental en el funcionamiento de la misma, pero constituyen una interacción poco estudiada.
No fue hasta 1978, cuando Kloepper and Schroth describieron a las rizobacterias promotoras del crecimiento vegetal o PGPR ( Plant growth-promoting rhizobacteria ) microorganismos que favorecen el buen desarrollo de las plantas. Estos pueden realizar muchas funciones, como solubilizar fosfatos, producir sideróforos, quitinasas, hormonas vegetales e incluso fijar nitrógeno. Y es este último dato el que más nos importa ahora mismo, ya que la simbiosis entre fijadores de nitrógeno y plantas extensamente cultivables supondría un tremendo ahorro en fertilizantes, además de beneficiar a los ecosistemas que se ven afectados por el exceso de residuos nitrogenados.
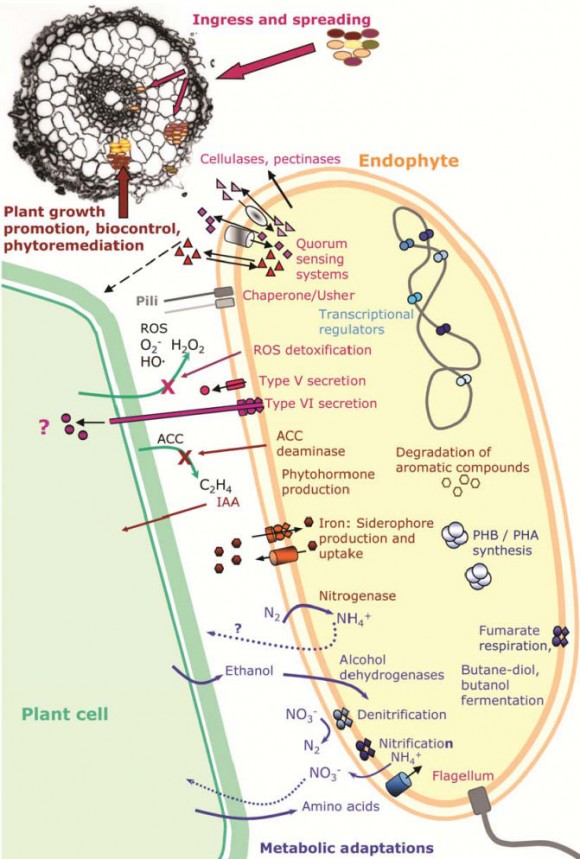
Como decía más arriba el análisis metagenómico consiste básicamente en mezclar la información genética de toda bacteria que viva en un determinado sitio, y luego analizarlo como si se tratase de un único organismo. Luego se comparan las secuencias con bases de datos buscando semejanzas con códigos conocidos. Imaginad que quisiéramos saber qué tipo de libros hay en una biblioteca, pero que la única forma que tuviésemos de verlos fuese pasándolos por una trituradora y guardando todos los restos mezclados, luego habría que unir trozos para hacerse una idea sobre el tipo de libros que existían allí.
Tras la secuenciación los investigadores encontraron genes codificantes para una multitud de proteínas, entre las que se encontraban una importante cantidad dedicada a la protección contra especies reactivas del oxígeno (ROS). A primera vista parece extraño, ya que las zonas donde se encuentran las raíces de plantas de arroz son terrenos inundados, y por ello, normalmente anóxicos. De hecho se esperaba encontrar una gran comunidad adaptada a la vida en un ambiente falto de oxígeno. Sin embargo el arroz posee un tejido llamado aerénquima que le permite mantener oxigenada su raíz, y con ello también a las bacterias que allí viven. También se encontraron genes que codificaban para proteínas implicadas en el metabolismo del nitrógeno y una gran cantidad de genes quorum sensing, implicados en funciones “sociales”.
Según parece las bacterias forman una comunidad organizada dentro de la raíz tapizando gran parte del tejido de la planta de forma análoga a lo que la flora intestinal hace con otros muchos animales, entre los que nos encontramos los humanos. Actuando sobre el propio tejido y sobre otras bacterias, sin embargo los análisis metagenómicos no nos permiten saber más, no nos ofrecen detalle de estas interacciones, para estudiar estas bacterias en profundidad hay que hacerlo de forma aislada, sembrando en placas y realzando todo tipo de experimentos.

La finalidad aplicada del estudio de los endofitos del arroz, persigue mejorar su crecimiento, abaratar su producción y salvaguardar el ecosistema de los fertilizantes. Para ello necesitamos conocer en la medida de lo posible las propiedades de cada bacteria, y cómo interaccionan entre sí, para así seleccionar aquellas que más beneficios aporten a la planta.
¿De qué beneficios hablamos?
Uno de los más buscados suele ser la fijación biológica de nitrógeno, conseguir bacterias que conviertan nitrógeno molecular en especies químicas asimilables para la planta es uno de los grandes atractivos de las PGPR. En plantas como la soja esto es ya una realidad que ahorra muchos millones al año. También buscamos bacterias que produzcan quitinasas para evitar el crecimiento de hongos, sideróforos que secuestran hierro, complicando la expansión de otros patógenos bacterianos, e incluso hormonas vegetales que aceleran el crecimiento de muchas plantas.
Para buscar estos beneficios necesitamos bacterias capaces de crecer en el laboratorio, y medios de cultivo que fomenten su crecimiento. Los medios no solo deben ser adecuados para el desarrollo y aislamiento de las PGPR, también tienen que potenciarlas frente a otros microorganismos.
Por ejemplo para el aislamiento de fijadores de nitrógeno usamos medios de cultivo que no contengan compuestos nitrogenados, de esta manera los microorganismos incapaces de obtener nitrógeno atmosférico (fijación biológica) serán incapaces de generar proteínas y material genético, siendo incapaces de crecer en las placas. Aunque la cosa no es tan simple, ya que muchos microorganismos no fijan nitrógeno en estado libre, otros necesitan la colaboración de segundas bacterias, sin olvidar a los que ni tan siquiera crecen en placas de cultivo.

Confirmada la fijación de nitrógeno, y realizadas otras muchas pruebas, deben realizarse distintos test con la planta objetivo, viendo si las mejoras esperadas son reales. Para ello se inoculan plantas con las distintas cepas bacterianas y se mantienen bajo condiciones controladas en las que se comprueban distintos parámetros como peso, tamaño, color… En el caso de la soja, las bacterias forman unos órganos llamados nódulos, dentro de los cuales se produce la fijación de nitrógeno. Los nódulos albergan a la bacteria alejada del exterior, y es la propia planta la que se encarga de llevar los nutrientes necesarios al nódulo alimentando a la bacteria para que realice la fijación de nitrógeno.

En el caso de el arroz, no se ha observado nunca la formación de nódulos. Sin embargo cuando inoculamos las plantas, crecidas en condiciones axénicas en medios semilíquidos carentes de nitrógeno combinado con algunas bacterias aisladas previamente de su rizosfera, en algunos casos aparecen extrañas formaciones en forma de campana presumiblemente compuestas por bacterias. Estás formaciones envuelven algunas zonas de la raíz. Quizás se trate de una nueva forma de simbiosis o quizás sólo sean agrupaciones de bacterias en los puntos donde las condiciones de desarrollo sean las óptimas, en el sentido de mayor concentración de exudados radicales y oxígeno. Sea lo que sean, el objetivo final de nuestro estudio es llegar a encontrar inóculos bacterianos del tipo de los conseguidos con los rizobios simbióticos para la soja, que permitan incrementar la producción de arroz eliminando los costes en fertilizantes y reduciendo el impacto de los mismos.
[youtube]http://www.youtube.com/watch?v=uyMFQNeAgFU[/youtube]
En este vídeo pueden observarse las misteriosas “campanas bacterianas” de distintos tamaños envolviendo a muchas de las raíces de la planta.
Quizás sea un proceso muy largo, pero no todo el mundo puede dedicarse a hacer calcetines…
———————————-
Referencias y agradecimientos:
Agradecimientos a M. R. Espuny por su ayuda y confianza; y a Manuel Bermudo por las fotos.
![]() Sessitsch A, Hardoim P, Döring J, Weilharter A, Krause A, Woyke T, Mitter B, Hauberg-Lotte L, Friedrich F, Rahalkar M, Hurek T, Sarkar A, Bodrossy L, van Overbeek L, Brar D, van Elsas JD, & Reinhold-Hurek B (2012). Functional characteristics of an endophyte community colonizing rice roots as revealed by metagenomic analysis. Molecular plant-microbe interactions : MPMI, 25 (1), 28-36 PMID: 21970692
Sessitsch A, Hardoim P, Döring J, Weilharter A, Krause A, Woyke T, Mitter B, Hauberg-Lotte L, Friedrich F, Rahalkar M, Hurek T, Sarkar A, Bodrossy L, van Overbeek L, Brar D, van Elsas JD, & Reinhold-Hurek B (2012). Functional characteristics of an endophyte community colonizing rice roots as revealed by metagenomic analysis. Molecular plant-microbe interactions : MPMI, 25 (1), 28-36 PMID: 21970692

José Jesús Gallego-Parrilla. Biólogo apasionado de la microbiología, actualmente haciendo el doctorado en la Universidad de Newcastle, en el grupo de Tracy Palmer. Escribo sobre bacterias en Microgaia.net y hablo un poco de todo en @Micro_Gaia. También me apasiona la ciencia ficción y todo lo relacionado con la tecnología.
