La vida es en gran parte intercambio y relación, ya seas un astronauta en la Estación Espacial Internacional o un porífero en el fondo del océano, tu supervivencia pasará por poder intercambiar gases y nutrientes con el medio que te rodea. Los microorganismos puede que dependan incluso de una forma más radical de este fenómeno de intercambio con el medio que les rodea, y es que si lo pensáis la inmensa mayoría de los procariotas no son más que células desnudas rodeadas de un universo de inclemencias y peligros. Imaginaos por un momento: sin ropa o piel que los protejan. Sin un sistema circulatorio o respiratorio que lleve nutrientes y energía allí donde hacen falta. Sin sistema nervioso que los avise de peligros a su alrededor… Ser un procariota no es desde luego nada fácil.
Básicamente y sin entrar en demasiado detalle, podemos decir que las células, no olvidemos que los procariotas no dejan de ser células, realizan transporte de forma activa o pasiva. En la forma pasiva tenemos difusión, ósmosis y difusión facilitada. En la forma activa podemos diferenciar entre endo y exocitosis según la molécula entre o salga de la célula. Al proceso de transporte o translocación de moléculas efectoras, como por ejemplo proteínas, enzimas o toxinas se le conoce como secreción. Estas moléculas realizan su función fuera de la célula, ya sea en el medio o en el espacio intermembrana que poseen las bacterias.
Aunque existen sistemas de transporte de membrana universales, por ejemplo los transportadores dependientes de ATP o transportadores ABC (del inglés ATP-binding cassette) ABC, que está presente en todos los reinos. Hoy quiero hablar de sistemas bacterianos principalmente. Como ya os he indicado arriba, los procariotas lo tienen mucho más difícil y por ello han acabado portando sistemas mucho más complejos… Así que os voy a presentar alguno de ellos antes de pararnos en el que más me interesa actualmente.
Sistema de secreción de tipo I (T1SS)
Estos sistemas los encontramos en bacterias Gram negativas y se encargan de transportar distintas moléculas incluyendo principalmente iones, carbohidratos, proteínas no plegadas y sobre todo toxinas de tipo RTX. El sistema está compuesto de un transportador ABC que se encuentra en la membrana interna, una proteína periplásmica que hace de túnel y una proteína tipo porina colocada en la membrana externa, recordad que las bacterias cuentan con un sistema de doble membrana.

Sistema de secreción tipo II (T2SS)
El T2SS es propio de bacterias Gram negativas y depende de dos sistemas de los que hablaremos más adelante: los sistemas Sec y Tat. Estos sistemas comienzan la secreción inicial hacia el espacio periplásmico, el cual se
encuentra entre la membrana interna y la externa. Una vez en el espacio periplasmático, la molécula transportada pasa a ser secretada por la parte externa del sistema que forma un canal de salida. Este componente externo está acompañado de otra proteína interna que asegura su estabilidad evitando que se degrade mientras realiza su trabajo, es la que podéis ver en azul en la figura. Las moléculas secretadas son principalmente enzimas implicadas en degradar otras membranas, por lo que este sistema tiene una importante relación con la capacidad de competir y la patogenia bacteriana.

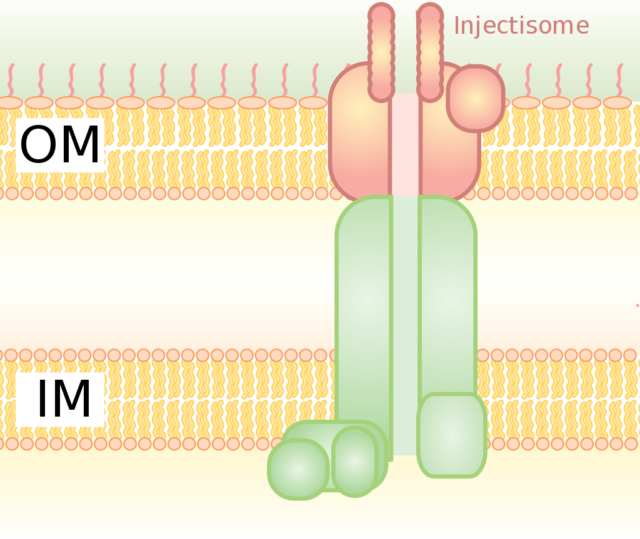
Sistema de secreción de tipo III (T3SS)
El T3SS es un sistema asociado al cuerpo basal del flagelo bacteriano, es propio de bacterias Gram negativas como Salmonella, Shigella, Yersinia o Vibrio entre otras. El T3SS es también conocido como inyectosoma porque se asemeja a una aguja que es capaz de transportar las moléculas
directamente desde el interior de las bacterias hacía el interior de otras células, convirtiéndose en un arma patogénica de gran capacidad.
Sistema de secreción de tipo IV (T4SS)
Este sistema está implicado en procesos de conjugación bacteriana, que vendría a ser algo así como la reproducción sexual bacteriana. Este sistema es capaz de transportar tanto ADN como proteínas no plegadas. Además de procesos de conjugación, también es responsable de introducir ADN en otros organismos, como en el caso de Agrobacterium tumefaciens del que hablé por encima aquí. Pero también es usado por otros famosos patógenos como Helicobacter pylori o Legionella pneumophila entre otros para secretar factores de virulencia. El mecanismo exacto por el que este sistema secreta o toma macromoléculas no es del todo conocido, pero es un sistema altamente versátil, capaz tanto de inyectar factores de virulencia o ADN en las células a las que ataca o de tomar ADN del medio que le rodea durante procesos de conjugación.

Sistema de secreción tipo V (T5SS)
Este sistema es conocido también como sistema de autotransporte y la razón, si lo pensáis, es bastante lógica. Casi todos los sistemas de secreción requieren energía para funcionar y transportar las moléculas a través de la membrana interna. Sin embargo en el periplasma, ese espacio que existe en las bacterias Gram negativas entre ambas membranas, la energía no está disponible. Esta ausencia de energía obliga a que algunas de las proteínas hayan encontrado formas de salir que no requieran ese chute de energía. Resumiendo y simplificando el proceso, podemos decir que gracias a los sistemas SEC o TAT, de los que ya hablaremos, la proteína es transportada de forma no plegada hasta el espacio periplasmático. Una vez en el espacio periplasmático, la proteína seguirá en estado no plegado protegida por chaperonas, y finalmente su inserción en la membrana será facilitada por la maquinaria del complejo BAM. Este sistema facilita el plegado y la inserción de proteínas en la membrana externa. Una vez integrada en la membrana será el sistema de secreción, formando un canal por el que secretará un trozo de ella misma. Podemos decir pues, que la proteína a secretar incorpora su propio sistema de secreción, de ahí el término autotransporte.

En la siguiente imagen podréis ver que existen varios subsistemas tipo V. El explicado es principalmente el tipo Va (autotransporte clásico). Los otros subtipos como el tipo Vb (o two-Partner Secretion) donde la proteína secretada no necesita ser “cortada” de la la proteína transportadora ya que forman dos unidades distintas. Casi todos los sistemas de secreción poseen subtipos, así que si os interesa os animo a leer la revisión de la bibliografía.

Sistema de transporte tipo VI (T6SS)
Este sistema fue descubierto no hace mucho (2006) en Vibrio choleare y Pseudomonas aeruginosa. La principal función de este sistema es la competencia bacteriana y la defensa frente a la predación por eucariontes unicelulares, algo sobre lo que ya escribí hace un tiempo aquí. Este sistema transporta proteínas desde el citoplasma de la bacteria hasta el interior de otra célula. EL T6SS está ampliamente extendido entre todo tipo de bacterias, desde patógenos de plantas y animales, bacterias marinas y bacterias propias del suelo. Aunque como decía la principal función de este sistema es la competencia entre bacterias. También es una herramienta usada contra células eucarióticas. Por ejemplo Francisella tularensis, agente causante de la tularemia usa su T6SS para sobrevivir y dividirse dentro de las células del sistema inmune que las intentan eliminar por medio de la fagocitosis.

Sistema de transporte tipo VII (T7SS)
El sistema de secreción tipo VII está presente en bacterias Gram positivas como por ejemplo Mycobacteria tuberculosis, M. bovis, Streptomyces coelicolor y Staphylococcus aureus entre otras. Como el T6SS el T7SS tiene dos roles principales: competición bacteriana y generar factores de virulencia contra eucariotas. El sistema produce dos toxinas principalmente EsaD que es una nucleasa (degrada ADN) y TspA una molécula que genera poros en la membrana de otras bacterias, aunque se sospecha que existen más toxinas. Es en general un sistema es bastante versátil y del que queda mucho por saber. Como además parte de mi grupo trabaja con él, seguramente le dedique una entrada en el futuro.

Sistema de transporte tipo VIII (T8SS)
El sistema de transporte tipo 8 funciona con la ruta nucleación-precipitación extracelular. Es propio de bacterias Gram negativas y se encarga de la secreción y ensamblaje de los pre-pilis para la biogénesis de las fimbrias/curli. Estas fimbrias son expansiones de la membrana celular a modo de pequeños “pelos” que las bacterias usan para adherirse a superficies o entre ellas. El T8SS sería pues la base que inicia la formación de estas estructuras

Sistema de transporte tipo IX (T9SS)
El sistema fue originalmente descrito en el filum Sphingobacteria, estas bacterias son por lo general algas unicelulares fotoautótrofas y anaeróbicas. El sistema está implicado en la movilidad bacteriana específicamente en el tipo gliding. Tras encontrar este sistema en el patógeno oral Porphyromonas gingivalis, también se vio que estaba implicado en algunos procesos patogénicos y en la degradación de biopolímeros.

Sistemas generales.
En la imagen superior podéis observar dos sistemas en la membrana interna en color violeta de los que no he hablado por ahora, la razón es que son de carácter general y a diferencia de los anteriores, estos pueden ser encontrados por lo general tanto en bacterias Gram negativas como en Gram positivas. Si tenéis curiosidad respecto a qué sistema corresponde a cada grupo, podéis acudir a esa revisión: (Green ER et al., 2016)
Sistema general de Secreción (Sec)
El sistema general de secreción es seguramente el sistema más conocido dentro del mundo de la microbiología. Ya que hasta finales de los 90 fue el único sistema general conocido y por ello ampliamente estudiado.
Este sistema, como los anteriores, se encarga de secretar proteínas no plegadas hacia el espacio periplásmico en las Gram negativas de doble membrana. Mientras que en las Gram positivas siguen otras rutas hacia el exterior celular. El sistema Sec utiliza dos rutas distintas: SecA y SRP de (signal recognition particle).
La vía de SecA permite que una proteína sea secretada casi directamente desde el ribosoma que la está produciendo. Parte del sistema además ayuda a que la secuencia proteica no se pliegue hasta que sea expulsada al periplasma mediante otra parte del sistema SecA que requiere energía. La vía SRP es similar con la diferencia de que esta vía integrará la secuencia proteica en la membrana interna.


He nombrado muchas veces el concepto de proteína no plegada, porque era quizás el punto central de esta historia. Las proteínas son inestables en el medio externo, el pH, la temperatura y otros factores pueden desnaturalizarlas y volverlas inútiles. Mientras estén dentro de la célula…están protegidas, pero fuera es otra historia totalmente distinta. Para los organismos pluricelulares esto es un problema menor, puesto que, si una de nuestras células secreta una proteína al medio extracelular, este seguirá siendo un lugar bastante estable en lo que a pH y temperatura se refiere. Pero imaginad que sois una bacteria en el suelo del bosque, luchando por hacerse por los recursos disponibles. Secretaría proteínas para atacar, para captar alimento o para defenderos. Todos los sistemas anteriores pueden ser de mucha utilidad, y eso fue lo que la comunidad científica pensó durante décadas…sin embargo había algo que no encajaba.

La mayoría de las proteínas se pliegan al salir de la membrana, ya sea por su propia estructura o por la ayuda de unas proteínas plegadoras llamadas chaperonas. Una vez plegada, la proteína es mucho más resistente y puede realizar su trabajo. Pongamos una metáfora:
Todos estos sistemas nombrados serían la versión bacteriana de Ikea, son capaces de exportar proteínas de la misma forma que Ikea vende muebles desmontados, y es que realmente a veces la única forma de meter en algunas casas muebles enormes es hacerlo por desmontados. Pero no todo lo que nos llega es desmontado. Siguiendo con la metáfora, que empieza a complicarse, a veces compramos cosas que por su complejidad no pueden ser montadas en casa, objetos muy caros y complejos como electrodomésticos. Pues igual existen proteínas que por contener cofactores metálicos y otras estructuras delicadas deben ser plegadas en el interior celular. ¡Nadie querría que le llegase una lavadora a casa desmontada tornillo a tornillo! Sin embargo, estás proteínas son enormes y hasta finales de los 90 no se supo como podrían ser exportadas sin comprometer la membrana bacteriana, un agujero del tamaño de dichas proteínas plegadas implicaría la rotura total y la muerte de la bacteria. Así que, ¿cómo pueden las bacterias secretar estas enormes proteínas?
La respuesta es… que aún no lo sabemos, ¡sin embargo ahora si que sabemos qué maquinaría utilizan!
Sistema Tat
El sistema Tat (twin arginine translocation system), es posiblemente el más misterioso de todos los sistemas de secreción, fue co-descubierto por Tracy Palmer y Ben Berks en el año 1998 y a día de hoy sigue siendo un auténtico misterio. Conocemos la estructura de los componentes del sistema, sabemos más o menos cómo se organizan…Pero a día de hoy el transporte y otras de sus funciones permanecen desconocidas.
Entre las diferencias entre el sistema Sec y Tat, además de la capacidad del segundo para exportar proteínas plegadas, podemos nombrar también otra, y es que está ausente en las células eucarióticas, siendo exclusivo de bacterias y arqueas. También podemos encontrarlo en algunos cloroplastos y en las mitocondrias de algunos organismos eucarióticos unicelulares. Pero como sistema completo es algo exclusivo del mundo procarionte, lo cual lo colocó desde el primer momento como una diana muy atractiva para la búsqueda de antibióticos. Y es que este sistema es esencial para la supervivencia en muchas bacterias patógenas y aunque no sea esencial para la supervivencia si que lo es para la virulencia en otras muchas bacterias.
Una de sus principales funciones es exportar proteínas implicadas en la remodelación de la membrana celular, muy implicado en la división y el crecimiento de la bacteria, algo que queda patente cuando lo eliminamos en bacterias donde no es vital, ya que estas
acaban creciendo de forma desorganizada generando largas cadenas.

Además de su influencia en la remodelación de la membrana, tiene otras funciones muy importantes, entre ellas: el metabolismo de fosfato y hierro, la resistencia a ciertos metales pesados, quorum sensing, respiración bacteriana en ausencia de oxígeno e incluso la simbiosis con plantas.
En Escherichia coli, una de las bacterias más estudiadas, se conocen más de 450 proteinas que utilizan la ruta Sec y tan sólo unas 30 que hagan uso de la ruta Tat, sin embargo como ya comentaba, estas casi 30 proteínas son de distintos tamaños y se exportan completamente plegadas, ¡sin afectar en absoluto la estabilidad de la membrana celular!
En E. coli encontramos tres componentes básicos para el sistema Tat: Tat A, Tat B y TatC. TatA y TatB son proteínas simples transmembrana, se parecen mucho entre ellas, lo que indica, espero hablaros de ello en el futuro, que provienen de un mismo ancestro. TatC es mucho mayor, posee 6 helices que cruzan la membrana y es el “andamio” sobre el que los otros componentes se reúnen para realizar el transporte de las proteínas.

Sabemos que existe probablemente un complejo en la membrana formado por los componentes ABC en igual proporción. Parece ser que cuando una proteína (sustrato) entra en contacto con TatC, se disparan una serie de modificaciones que desencadenan en el reclutamiento de muchas unidades de TatA que de alguna forma transportan el sustrato al exterior de la membrana interna.

Como podéis ver, el mundo de la secreción bacteriana es aún tierra por descubrir, un lugar lleno de misterios, este texto es una pequeña introducción, quizás simplista para los más expertos, pero que espero os haya servido para conocer un poco mejor cómo los seres invisibles se las arreglan para relacionarse con su entorno.
Y quién sabe, quizás con un poco de suerte, ¡pueda volver pronto a contaros novedades sobre algunos de estos sistemas!
Fuentes y links para ampliar:
Linhartová, Irena; Bumba, Ladislav; Mašín, Jiří; Basler, Marek; Osička, Radim; Kamanová, Jana; Procházková, Kateřina; Adkins, Irena; Hejnová-Holubová, Jana (2010-11-01) «RTX proteins: a highly diverse family secreted by a common mechanism». FEMS Microbiology Reviews. 34 (6): 1076–1112. doi:10.1111/j.1574-6976.2010.00231.x. ISSN 1574-6976. PMC 3034196.. PMID 20528947.
Thanassi, D. G., Stathopoulos, C., Karkal, A., & Li, H. (2005). Protein secretion in the absence of ATP: the autotransporter, two-partner secretion and chaperone/usher pathways of Gram-negative bacteria (Review). Molecular Membrane Biology, 22(1-2), 63–72. doi:10.1080/09687860500063290
https://www.cell.com/structure/pdfExtended/S0969-2126(17)30010-2
Douzi B, Filloux A, Voulhoux R (2012). «On the path to uncover the bacterial type II secretion system». Philosophical Transactions of the Royal Society B: Biological Sciences. 367: 1059–1072. doi:10.1098/rstb.2011.0204. PMC 3297435. PMID 22411978.
https://www.medigraphic.com/pdfs/revedubio/reb-2011/reb112b.pdf
MacIntyre DL, Miyata ST, Kitaoka M, & Pukatzki S (2010). The Vibrio cholerae type VI secretion system displays antimicrobial properties. Proceedings of the National Academy of Sciences of the United States of America, 107 (45), 19520-4 PMID: 20974937
AgfC and AgfE facilitate extracellular thin aggregative fimbriae synthesis in Salmonella enteritidis. Gibson DL, White AP, Rajotte CM, Kay WW. Microbiology (Reading, Engl.) 153, 1131-40, (2007). PMID: 17379722
Veith PD, Glew MD, Gorasia DG, Reynolds EC (October 2017). «Type IX secretion: the generation of bacterial cell surface coatings involved in virulence, gliding motility and the degradation of complex biopolymers». Molecular Microbiology. 106 (1): 35–53. doi:10.1111/mmi.13752.
Green ER, Mecsas J (February 2016). Kudva IT (ed.). «Bacterial Secretion Systems: An Overview». Microbiology Spectrum (5 ed.). American Society for Microbiology Press. 4 (1): 215–239.
The Twin-arginine translocation (Tat) system is essential for Rhizobium-legume symbiosis July 2003 Molecular Microbiology 48(5):1195-207 DOI: 10.1046/j.1365-2958.2003.03510.x
Palmer, T., & Stansfeld, P. J. (2020). Targeting of proteins to the twin‐arginine transport pathway. Molecular Microbiology. doi:10.1111/mmi.14461
Han, L., Zheng, J., Wang, Y., Yang, X., Liu, Y., Sun, C., … Huang, Y. (2016). Structure of the BAM complex and its implications for biogenesis of outer-membrane proteins. Nature Structural & Molecular Biology, 23(3), 192–196. doi:10.1038/nsmb.3181
Meuskens, I., Saragliadis, A., Leo, J. C., & Linke, D. (2019). Type V Secretion Systems: An Overview of Passenger Domain Functions. Frontiers in Microbiology, 10. doi:10.3389/fmicb.2019.01163

José Jesús Gallego-Parrilla. Biólogo apasionado de la microbiología, actualmente haciendo el doctorado en la Universidad de Newcastle, en el grupo de Tracy Palmer. Escribo sobre bacterias en Microgaia.net y hablo un poco de todo en @Micro_Gaia. También me apasiona la ciencia ficción y todo lo relacionado con la tecnología.
